
微生物+单细胞分选仪 拉曼单细胞分选仪 菌落原位多表型检测与挑取工作站 单细胞光镊操纵与分选系统 |
拉曼+共聚焦拉曼光谱仪 近红外共聚焦拉曼光谱仪 拉曼单细胞分选仪 |
成像+超快三维荧光成像系统 智能细胞荧光计数仪制药颗粒物检测仪 |
核酸快检便携式核酸恒温扩增分析仪耗材拉曼信号增强芯片 细胞分选芯片 |

微生物+单细胞分选仪 拉曼单细胞分选仪 菌落原位多表型检测与挑取工作站 单细胞光镊操纵与分选系统 |
拉曼+共聚焦拉曼光谱仪 近红外共聚焦拉曼光谱仪 拉曼单细胞分选仪 |
成像+超快三维荧光成像系统 智能细胞荧光计数仪制药颗粒物检测仪 |
核酸快检便携式核酸恒温扩增分析仪耗材拉曼信号增强芯片 细胞分选芯片 |
2022年9月26日,中国科学院城市环境研究所朱永官院士、崔丽研究员团队应用长光辰英核心产品—PRECI SCS单细胞分选仪在《PNAS》期刊上发表了题为“Active antibiotic resistome in soils unraveled by single-cell isotope probing and targeted metagenomics”的论文,文章第一作者为李弘哲博士,该文章通过发展单细胞拉曼-稳定同位素标记和靶向宏基因组联用技术,示踪了土壤原位活性抗生素耐药菌,量化了其表型耐药水平,并结合单细胞靶向分选与测序揭示了土壤高活性耐药菌的抗性组和移动组。

土壤中的抗生素抗药性(AMR)通过食物链方式转移至人类而带来健康威胁。活性耐药菌在驱动土壤耐药性传播中起着关键作用,然而,针对土壤原位活性耐药菌(ARB)的探索仍很少。本研究通过发展单细胞拉曼-稳定同位素标记和靶向宏基因组联用技术,示踪了土壤原位活性抗生素耐药菌,量化了其表型耐药水平,并结合单细胞靶向分选与测序揭示了土壤高活性耐药菌的抗性组和移动组。科研团队在单细胞水平破译了活性耐药菌携带的抗性基因、毒力因子、可移动遗传元件(包括质粒、插入序列和前噬菌体)。该工作将多种抗生素耐药表型和多种基因型关联,为剖析环境中大量未培养耐药菌提供了崭新的方法。
本研究利用单细胞拉曼-重水同位素标记技术,针对土壤的复杂性以及对抗生素有效性的影响,通过优化抗生素剂量、孵育时间、采谱深度,建立了准确示踪土壤活性耐药菌的单细胞方法与判别标准,利用土壤原位环境的多种已知抗性菌和敏感菌,对方法在不同土壤和不同机制抗生素的普适性和准确性进行交互验证,将方法从简单的临床耐药菌的研究拓展至包含大量未培养菌的复杂土壤环境。本研究针对拉曼技术识别具有潜在健康风险的高度活跃土壤耐药菌,利用单细胞分选与靶向宏基因组测序技术,鉴定出多数高表型耐药菌属于之前难以研究的未培养菌以及一株新型的抗生素抗性病原菌,证明了土壤未培养菌是AMR的重要宿主。科研团队在单细胞水平破译了活性耐药菌携带的抗性基因、毒力因子、可移动遗传元件(包括质粒、插入序列和前噬菌体)。该工作将多种抗生素耐药表型和多种基因型关联,为剖析环境中大量未培养耐药菌提供了崭新的方法。
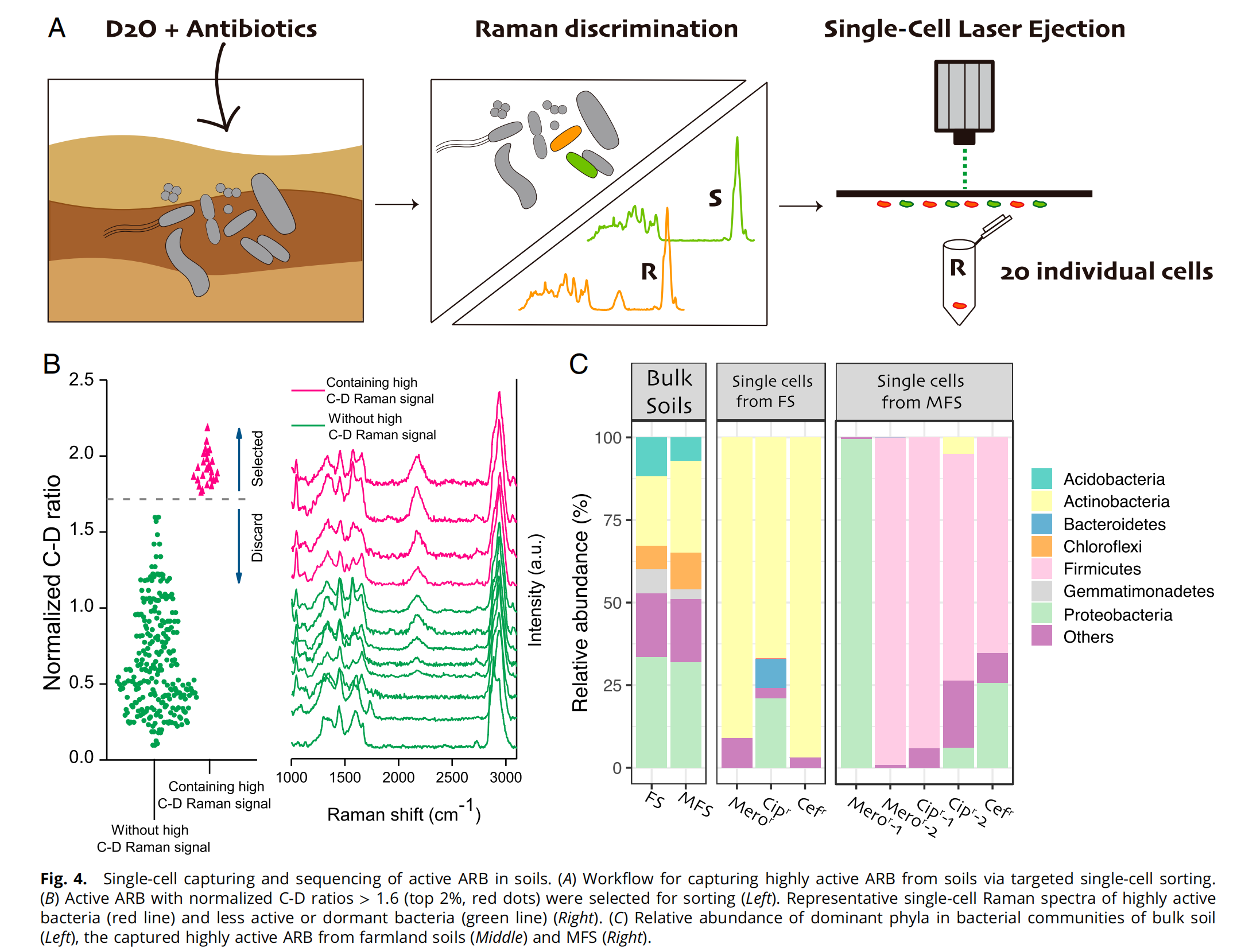
本研究利用单细胞拉曼-重水同位素标记技术,并且结合单细胞靶向分选与测序技术,在单细胞水平和表型层面克服培养限制,直接示踪和定量了土壤原位活性耐药菌的丰度和活性水平,揭示了人类活动显著增加土壤的表型耐药水平。该工作研究为复杂环境耐药研究提供了新手段,深化了科学家对土壤活性抗生素耐药性的认知。该方法可广泛用于其他生态系统,并对在“One Health”框架下推进环境耐药性的风险评估与制定防控策略具有重要价值。
原文链接:
https://www.pnas.org/doi/10.1073/pnas.2201473119


 0431-81077008
0431-81077008